NEWS
NEWS
Comb jellies reveal ancient origins of animal genome regulation
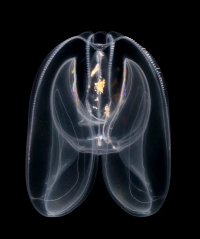
Mnemiopsis leidyi, one of the comb jellies studied which helped reveal the origins of distal regulation. Credit: Joan-Josep Soto Angel
Life depends on genes being switched on and off at exactly the right time. Even the simplest living organisms do this, but usually over short distances across the DNA sequence, with the on/off switch typically right next to a gene. This basic form of genomic regulation is probably as old as life on Earth.
A new study published today in Nature by researchers at the Centre for Genomic Regulation (CRG) and the Centre Nacional d’Anàlisi Genòmica (CNAG) finds that the ability to control genes from far away, over many tens of thousands of DNA letters, evolved between 650 and 700 million years ago. It probably appeared at the very dawn of animal evolution, around 150 million years earlier than previously thought.
Long-distance gene control, called distal regulation, relies on physically folding DNA and proteins into sophisticated loops. This allows regions far from a gene’s starting point to activate its function. This extra layer of control likely helped the first multicellular animals build specialised cell types and tissues without inventing new genes.
The critical innovation likely originated in a sea creature, the common ancestor or all extant animals. The ancient animal evolved the ability to fold DNA in a controlled manner, creating loops in three-dimensional space that brought far-flung bits of DNA in direct contact with each other.
“This creature could repurpose its genetic toolkit in different ways like a Swiss knife, enabling it to refine and explore innovative survival strategies. We did not expect this layer of complexity to be so ancient,” says Dr. Iana Kim, first author of the study and postdoctoral researcher with dual affiliation between the Centre for Genomic Regulation (CRG) and the Centre Nacional d’Anàlisis Genòmica (CNAG).
The authors of the study made the discovery by exploring the genomes of many of the oldest branches on the animal family tree, including comb jellies like the ‘sea walnut’ (Mnemiopsis leidyi), placozoans, cnidarians, and sponges. They also studied single-celled relatives that are not animals but share a recent common ancestor.
“You can discover a lot of new biology by looking at weird sea creatures. So far, we had been comparing genome sequences, but thanks to new methods we can now analyse which gene regulation mechanisms control genome function across species,” explains ICREA Research Professor Arnau Sebe-Pedrós, corresponding author of the study and Group Leader at the Centre for Genomic Regulation.
The team used a technique called Micro-C to map how DNA physically folds inside the cells of each of the 11 different species they studied. For scale, each human cell nucleus packs about two metres of DNA. The researchers sifted through 10 billion pieces of sequencing data to build each species’ 3D genome map in detail.
While there was no evidence of distal regulation in the single-celled relatives of animals, early-branching animals like comb jellies, placozoans and cnidarians had many loops. The sea walnut alone had over four thousand loops genome-wide. The finding is surprising given its genome is around just 200 million DNA letters long. In comparison, the human genome is 3.1 billion letters long and our cells can have tens of thousands of loops.
Until now, distal regulation was thought to have first appeared in the last common ancestor of bilaterians, a group of many different types of animals which first appeared on Earth around 500 million years ago. However, comb jellies are descended from life forms which diverged early from other animal lineages around 650 to 700 million years ago.
Whether comb jellies are older than sponges in the tree of life is a longstanding debate in evolutionary biology circles, but the study demonstrates that distal regulation arose at least one hundred and fifty million years earlier than previously thought.
The study made another surprising discovery. Many animals are vertebrates. In their cells, loops are controlled by CTCF, an architectural protein which defines boundaries and compartmentalises genes into different local neighbourhoods. It is a foundational unit of genomic architecture in mammals, birds, reptiles, amphibians, and fish. However, the genomes of the early-branching animals do not encode any equivalent protein to CTCF. Instead, the authors discovered that comb jellies use a different architectural protein belonging to the same structural family. The discovery shatters the assumption that advanced genomic, distal regulation require CTCF.
“It is impressive that the same problem has been solved using different tools. Thanks to this work, we now know that you can use two different proteins to bring distal DNA pices together in space forming a loop. Isn’t evolution marvellous?” says ICREA Research Professor Marc A. Marti-Renom, Group Leader at the Centre Nacional d’Anàlisi Genomic and the Centre for Genomic Regulation.
Like sponges and comb jellies, humans are also made of the same building blocks of DNA. Today, our bodies rely on the ancient innovation of distal regulation to help create different types of cells from the same DNA, producing everything from brain cells to immune cells. When these contacts go wrong, diseases can arise.
By tracing distal regulation to animals that lived many hundreds of million years ago, researchers can begin to piece together how the earliest versions of genomic regulation took shape, providing new clues about the fundamental principles that govern our cells and bodies today. This can help us understand where the system is robust and where it’s prone to failure, potentially guiding new medical insights or therapies.
This work was led by the Centre for Genomic Regulation (CRG), in collaboration with Marc A. Marti Renom’s Group at the Centre Nacional d’Anàlisis Genòmica, the University of Bergen, Queen Mary University of London, the Prefectural University of Hiroshima, and the University of Alberta. It was funded by an ERC Starting Grant (European Research Council) from the European Union.
EN CASTELLANO
Las medusas peine revelan el origen primitivo de la regulación genómica de los animales
La vida depende de la activación y la desactivación de los genes en un momento específico. Esto ocurre incluso en los seres vivos más simples, pero generalmente en distancias cortas a través de la secuencia de ADN, con el interruptor de encendido o apagado colocado justo al lado de un gen. Este tipo de regulación genómica es básico y probablemente tan antiguo como la vida en la Tierra.
Un nuevo estudio publicado hoy en Nature por un equipo del Centro de Regulación Genómica (CRG) y del Centro Nacional de Análisis Genómico (CNAG) ha descubierto que la capacidad de controlar genes desde más lejos, a través de muchas decenas de miles de letras de ADN, evolucionó hace entre 650 y 700 millones de años. Probablemente apareció en los inicios de la evolución de los animales, unos 150 millones de años antes de lo que se creía.
El control genético a larga distancia, o regulación distal, se basa en el plegamiento físico del ADN y las proteínas en bucles sofisticados. Esto permite que regiones alejadas del punto de partida de un gen activen su función. Es una capa adicional de control que probablemente ayudó a los primeros animales multicelulares a construir tipos de células y tejidos especializados sin tener que inventar nuevos genes.
Es probable que la innovación crítica se originara en una criatura marina, en el ancestro común de todos los animales existentes. El animal desarrolló la capacidad de plegar el ADN de manera controlada, creando bucles en el espacio tridimensional que ponían en contacto directo fragmentos de ADN lejanos entre sí en un plano bidimensional.
"Esta criatura podría reutilizar su conjunto de herramientas genéticas de diferentes maneras de forma similar a una navaja suiza, lo que le permitiría refinar y explorar estrategias de supervivencia innovadoras. No esperábamos que esta capa de complejidad fuera tan antigua", afirma la Dra. Iana Kim, coautora del estudio e investigadora postdoctoral con doble afiliación entre el Centro de Regulación Genómica (CRG) y el Centro Nacional de Análisis Genómico (CNAG).
El descubrimiento fue posible gracias a la exploración de los genomas de muchas de las ramas más antiguas del árbol genealógico de los animales, incluidas las medusas peine como la "nuez de mar" (Mnemiopsis leidyi), los placozoos, los cnidarios y las esponjas. También se estudiaron parientes unicelulares que no son animales, pero que comparten un ancestro común.
"Se puede descubrir una gran cantidad de biología nueva observando extrañas criaturas marinas. Hasta ahora, habíamos estado comparando secuencias genómicas, pero gracias a nuevos métodos ahora podemos analizar qué mecanismos de regulación génica controlan la función del genoma en todas las especies", explica el profesor de investigación ICREA Arnau Sebe Pedrós, coautor principal del estudio y líder del grupo en el Centro de Regulación Genómica.
El equipo utilizó una técnica llamada Micro-C para mapear cómo el ADN se pliega físicamente dentro de las células de cada una de las 11 especies diferentes que estudiaron. A escala, cada núcleo de célula humana contiene unos dos metros de ADN. Examinaron 10.000 millones de datos de secuenciación para construir el mapa del genoma en 3D de cada especie en detalle.
Si bien no hubo evidencia de regulación distal en los parientes unicelulares de los animales, los animales no bilaterales, como las medusas peine, los placozoos y los cnidarios, tenían muchos bucles. Mnemiopsis leidyi tenía más de cuatro mil bucles en todo el genoma. El hallazgo es sorprendente dado que su genoma tiene alrededor de 200 millones de letras de ADN. En comparación, el genoma humano tiene 3.100 millones de letras y nuestras células pueden tener decenas de miles de bucles.
Hasta ahora, se pensaba que la regulación distal había aparecido por primera vez en el último ancestro común de los animales bilaterales, un grupo de muchos tipos diferentes de animales que apareció por primera vez en la Tierra hace unos 500 millones de años. Sin embargo, las medusas peine descienden de formas de vida que divergieron temprano de otros linajes de animales hace unos 650 o 700 millones de años.
Que las medusas peine sean más antiguas que las esponjas en el árbol de la vida sigue siendo un debate en círculos de biología evolutiva, pero el estudio demuestra que la regulación distal surgió al menos ciento cincuenta millones de años antes de lo que se creía.
El estudio hizo otro descubrimiento sorprendente. Muchos animales son vertebrados. En sus células, los bucles están controlados por CTCF, una proteína arquitectónica que define los límites y compartimenta los genes en diferentes vecindarios locales. Es una unidad fundamental de la arquitectura genómica en mamíferos, aves, reptiles, anfibios y peces.
Sin embargo, los genomas de los animales no bilaterales no codifican ninguna proteína equivalente a la CTCF. En cambio, los autores descubrieron que las medusas peine usan una proteína arquitectónica diferente que pertenece a la misma familia estructural. El descubrimiento rompe la suposición de que la regulación genómica distal avanzada requiere CTCF.
"Es impresionante que el mismo problema se haya resuelto con diferentes herramientas. Gracias a este trabajo, ahora sabemos que se pueden utilizar dos proteínas diferentes para unir las piezas distales de ADN en el espacio formando un bucle. ¿No es maravillosa la evolución?", afirma el profesor de investigación ICREA Marc A. Marti-Renom, jefe de grupo del Centro Nacional de Análisis Genómico y del Centro de Regulación Genómica.
Al igual que las esponjas y las medusas peine, los humanos también están hechos de los mismos componentes básicos del ADN. Hoy en día, nuestros cuerpos dependen de la antigua innovación de la regulación distal para ayudar a crear diferentes tipos de células a partir del mismo ADN, produciendo de todo, desde células cerebrales hasta células inmunitarias. Cuando estos contactos no funcionan, pueden surgir enfermedades.
Al rastrear la regulación distal hasta animales que vivieron hace muchos cientos de millones de años, se puede comenzar a reconstruir cómo tomaron forma las primeras versiones de la regulación genómica, proporcionando nuevas pistas sobre los principios fundamentales que gobiernan nuestras células y cuerpos en la actualidad. Esto puede ayudarnos a comprender en qué puntos el sistema es sólido y dónde es propenso a fallar, lo que podría guiar nuevos conocimientos o terapias médicas.
El trabajo ha sido liderado por el Centro de Regulación Genómica (CRG), en colaboración con el grupo de Marc A. Martí Renom en el Centro Nacional de Análisis Genómico, la Universidad de Bergen, la Universidad Queen Mary de Londres, la Universidad de la Prefectura de Hiroshima y la Universidad de Alberta; y se ha financiado con una ayuda ERC Starting Grant del Consejo Europeo de Investigación de la Unión Europea.
EN CATALÀ
Les nous de mar revelen l'origen primitiu de la regulació genòmica dels animals
La vida depèn de l'activació i la desactivació dels gens en un moment específic. Això passa en els éssers vius més simples, però generalment en distàncies curtes a través de la seqüència d'ADN, amb l'interruptor d'encesa o apagat col·locat just al costat d'un gen. Aquest tipus de regulació genòmica és bàsic i probablement tan antic com la vida a la Terra.
Un nou estudi publicat avui a Nature per un equip del Centre de Regulació Genòmica (CRG) i del Centre Nacional d'Anàlisi Genòmica (CNAG) ha descobert que la capacitat de controlar gens des de més lluny, a través de moltes desenes de milers de lletres d'ADN, va evolucionar fa entre 650 i 700 milions d'anys. Probablement va aparèixer en els inicis de l'evolució dels animals, uns 150 milions d'anys abans del que es creia.
El control genètic a llarga distància, o regulació distal, es basa en el plegament físic de l'ADN i les proteïnes en bucles sofisticats. Això permet que regions allunyades del punt de partida d'un gen activin la seva funció. És una capa addicional de control que probablement va ajudar els primers animals multicel·lulars a construir tipus de cèl·lules i teixits especialitzats sense haver d'inventar nous gens.
És probable que la innovació crítica s'originés en una criatura marina, en l'ancestre comú de tots els animals existents. L'animal va desenvolupar la capacitat de plegar l'ADN de manera controlada, creant bucles en l'espai tridimensional que posaven en contacte directe fragments d'ADN llunyans entre si en un pla bidimensional.
"Aquesta criatura podria reutilitzar el seu conjunt d'eines genètiques de diferents maneres de forma similar a una navalla suïssa, fet que li permetria refinar i explorar estratègies de supervivència innovadores. No esperàvem que aquesta capa de complexitat fos tan antiga", afirma la Dra. Iana Kim, coautora de l'estudi i investigadora postdoctoral amb doble afiliació entre el Centre de Regulació Genòmica (CRG) i el Centre Nacional d'Anàlisi Genòmica (CNAG).
El descobriment va ser possible gràcies a l'exploració dels genomes de moltes de les branques més antigues de l'arbre genealògic dels animals, incloses les les "nous de mar" (Mnemiopsis leidyi), els placozous, els cnidaris i les esponges. També es van estudiar parents unicel·lulars que no són animals, però que comparteixen un ancestre comú.
"Es pot descobrir una gran quantitat de biologia nova observant estranyes criatures marines. Fins ara, havíem estat comparant seqüències genòmiques, però gràcies a nous mètodes ara podem analitzar quins mecanismes de regulació gènica controlen la funció del genoma en totes les espècies", explica el Professor d’Investigació ICREA Arnau Sebe Pedrós, coautor principal de l'estudi i líder del grup al Centre de Regulació Genòmica.
L'equip va utilitzar una tècnica anomenada Micro-C per observar com l'ADN es plega físicament dins de les cèl·lules de cadascuna de les 11 espècies diferents que van estudiar. A escala, cada nucli de cèl·lula humana conté uns dos metres d'ADN. Van examinar 10.000 milions de dades de seqüenciació per construir el mapa del genoma en 3D de cada espècie en detall.
Si bé no hi va haver evidència de regulació distal en els parents unicel·lulars dels animals, els animals no bilaterals, com les ‘nous de mar’, els placozous i els cnidaris, tenien molts bucles. Mnemiopsis leidyi tenia més de quatre mil bucles en tot el genoma. La troballa és sorprenent atès que el seu genoma té al voltant de 200 milions de lletres d'ADN. En comparació, el genoma humà té 3.100 milions de lletres i les nostres cèl·lules poden tenir desenes de milers de bucles.
Fins ara, es pensava que la regulació distal havia aparegut per primera vegada en l'últim ancestre comú dels animals bilaterals, un grup de molts tipus diferents d'animals que va aparèixer per primera vegada a la Terra fa uns 500 milions d'anys. No obstant això, les ‘nous de de mar’ descendeixen de formes de vida que van divergir aviat d'altres llinatges d'animals fa uns 650 o 700 milions d'anys.
Que les ‘nous de mar’ siguin més antigues que les esponges a l'arbre de la vida continua essent un debat en cercles de biologia evolutiva, però l'estudi demostra que la regulació distal va sorgir almenys cent cinquanta milions d'anys abans del que es creia.
L'estudi va fer un altre descobriment sorprenent. Molts animals són vertebrats. En les seves cèl·lules, els bucles estan controlats per CTCF, una proteïna arquitectònica que defineix els límits i compartimenta els gens en diferents veïnats locals. És una unitat fonamental de l'arquitectura genòmica en mamífers, aus, rèptils, amfibis i peixos.
No obstant això, els genomes dels animals no bilaterals no codifiquen cap proteïna equivalent a la CTCF. En canvi, els autors van descobrir que les ‘nous de mar’ fan servir una proteïna arquitectònica diferent que pertany a la mateixa família estructural. El descobriment trenca la suposició que la regulació genòmica distal avançada requereix CTCF.
"És impressionant que el mateix problema s'hagi resolt amb diferents eines. Gràcies a aquest treball, ara sabem que es poden utilitzar dues proteïnes diferents per unir les peces distals d'ADN a l'espai formant un bucle. No és meravellosa l'evolució?", afirma el Professor d’Investigació ICREA Marc A. Marti-Renom, cap de grup del Centre Nacional d'Anàlisi Genòmica i del Centre de Regulació Genòmica.
Igual que les esponges i les ‘nous de mar’, els humans també estan fets dels mateixos components bàsics de l'ADN. Avui dia, els nostres cossos depenen de l'antiga innovació de la regulació distal per ajudar a crear diferents tipus de cèl·lules a partir del mateix ADN, produint de tot, des de cèl·lules cerebrals fins a cèl·lules immunitàries. Quan aquests contactes no funcionen, poden sorgir les malalties.
En rastrejar la regulació distal fins a animals que van viure fa molts centenars de milions d'anys, es pot començar a reconstruir la manera en què van prendre forma les primeres versions de la regulació genòmica, proporcionant noves pistes sobre els principis fonamentals que governen les nostres cèl·lules i cossos en l'actualitat. Això pot ajudar-nos a comprendre en quins punts el sistema és sòlid i on és propens a fallar, cosa que podria guiar nous coneixements o teràpies mèdiques.
El treball ha estat liderat pel Centre de Regulació Genòmica (CRG), en col·laboració amb el grup de Marc A. Martí Renom al Centre Nacional de Anàlisi Genòmica, la Universitat de Bergen, la Universitat Queen Mary de Londres, la Universitat de la Prefectura d'Hiroshima i la Universitat d'Alberta; i s'ha finançat amb un ajut ERC Starting Grant del Consell Europeu de Recerca de la Unió Europea.