NOTÍCIES
NOTÍCIES
Abundant ‘secret doors’ on human proteins could reshape drug discovery
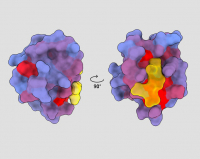
A three-dimensional image showing the human protein PSD95-PDZ3 from different angles. A molecule binding to the active site shown in yellow, with the blue-to-red colour gradient indicating increasing potential for allosteric effects. Based on PDB accession 1BE9. Credit: André Faure/ChimeraX
The number of potential therapeutic targets on the surfaces of human proteins is much greater than previously thought, according to the findings of a new study in the journal Nature.
A ground-breaking new technique developed by researchers at the Centre for Genomic Regulation (CRG) in Barcelona has revealed the existence of a multitude of previously secret doors that control protein function which could, in theory, be targeted to dramatically change the course of diseases as varied as dementia, cancer and infectious diseases.
The method, in which tens of thousands of experiments are performed at the same time, has been used to chart the first ever map of these elusive targets, also known as allosteric sites, in two of the most common human proteins, revealing they are abundant and identifiable.
The approach could be a game changer for drug discovery, leading to safer, smarter and more effective medicines. It enables research labs around the world to find and exploit vulnerabilities in any protein – including those previously thought ‘undruggable’.
“Not only are these potential therapeutic sites abundant, there is evidence they can be manipulated in many different ways. Rather than simply switching them on or off, we could modulate their activity like a thermostat. From an engineering perspective, that’s striking gold because it gives us plenty of space to design ‘smart drugs’ that target the bad and spare the good,” explains André Faure, postdoctoral researcher at the CRG and co-first author of the paper.
Proteins play a central role in all living organisms and carry out vital functions such as providing structure, speeding up reactions, acting as messengers or fighting disease. They are made of amino acids, folding into countless different shapes in three-dimensional space. The shape of a protein is crucial for its function, with just one mistake in an amino acid sequence resulting in potentially devastating consequences for human health.
Allostery is one of the great unsolved mysteries of protein function. Allosteric effects occur when a molecule binds to the surface of a protein, which in turn causes changes at a distant site in the same protein, regulating its function by remote control. Many disease-causing mutations, including numerous cancer drivers, are pathological because of their allosteric effects.
Despite their fundamental importance, allosteric sites are incredibly difficult to find. This is because the rules governing how proteins work at the atomic level are hidden out of sight. For example, a protein might shapeshift in the presence of an incoming molecule, revealing hidden pockets deep within its surface that are potentially allosteric but not identifiable using conventional structure determination alone.
Drug hunters have traditionally designed treatments that target a protein’s active site, the small region where chemical reactions occur or targets are bound. The downside of these drugs, also known as orthosteric drugs, is that active sites of many proteins look very similar and so drugs tend to bind and inhibit many different proteins at once, leading to potential side effects. In comparison, the specificity of allosteric sites means that allosteric drugs are some of the most effective types of medication currently available. Many allosteric drugs, which treat various conditions ranging from cancer to AIDS to hormone disorders, have been discovered by accident.
The authors of the study addressed this challenge by developing a technique called double deep PCA (ddPCA), which they describe as a ‘brute force experiment’. “We purposefully break things in thousands of different ways to build a complete picture of how something works”, explains ICREA Research Professor Ben Lehner, Coordinator of the Systems Biology programme at the CRG and author of the study. “It’s like suspecting a faulty spark plug, but instead of only checking that, the mechanic dismantles the entire car and checks it piece by piece. By testing ten thousand things in one go we identify all the pieces that really matter.”
The method works by changing the amino acids that make up a protein, resulting in thousands of different versions of the protein with just one or two differences in the sequence. The effects of the mutations are then tested all at the same time in living cells in the laboratory.
“Each cell is a tiny factory making a different version of the protein. In a single test tube we have millions of different factories and so we can very rapidly test how well all the different versions of a protein work”, adds Dr. Lehner. The data collected from the experiments is fed into neural networks, algorithms that analyze data by mimicking the way the human brain operates, which result in comprehensive maps that pinpoint the location of allosteric sites on the surfaces of proteins.
One of the great advantages of the method is that it is an affordable technique accessible to any research lab around the world. “It massively simplifies the process needed to find allosteric sites, with the technique working at a level of accuracy better than several different more expensive and time-consuming lab methods”, says Júlia Domingo, co-first author of the study. “Our hope is that other scientists use the technique to rapidly and comprehensively map the allosteric sites of human proteins one by one.”
One of the longer-term benefits of the technique is its potential to study the function and evolution of proteins. The authors of the study believe that, if scaled up, the method could one day result in advances that can precisely predict the properties of proteins from their amino acid sequences. If successful, the authors argue this would usher in a new era of predictive molecular biology, allowing much faster development of new medicine and clean, biology-based industry.
“While some tools can predict a protein’s structure by reading its sequence, our method goes one step further by telling us how a protein works. This is part of a bigger vision to make biology as engineerable as aeroplanes, bridges or computers. We have faced the same challenges for over 70 years, but it turns out they are more tractable than we previously thought. If we succeed it will open a new field with unprecedented possibilities”, concludes Dr. Lehner.
EN CASTELLANO
Una multitud de ‘puertas ocultas’ en las proteínas humanas podría transformar el descubrimiento de fármacos
El número de posibles dianas terapéuticas que hay en la superficie de las proteínas humanas es mucho mayor de lo que se creía, según los resultados de un nuevo estudio publicado en la revista Nature.
Una nueva técnica innovadora desarrollada por un equipo científico del Centro de Regulación Genómica (CRG) de Barcelona ha revelado la existencia de multitud de ‘puertas ocultas’ que controlan la función de las proteínas y que podrían, en teoría, utilizarse como dianas para cambiar drásticamente la evolución de enfermedades tan diversas como la demencia, el cáncer y las enfermedades infecciosas.
El método, en el que se ejecutan decenas de miles de experimentos a la vez, se ha utilizado para trazar el primer mapa de estas dianas tan difíciles de encontrar, conocidas como sitios alostéricos, en dos de las proteínas humanas más habituales, lo cual ha evidenciado que son abundantes e identificables.
Esta técnica podría cambiar las normas del juego en el descubrimiento de fármacos y dar lugar a unos medicamentos más seguros, más inteligentes y más eficaces. Permite a los laboratorios de investigación de todo el mundo buscar y aprovechar las vulnerabilidades de cualquier proteína, incluidas las que hasta ahora se consideraban «dianas imposibles».
«No solo resulta que estos potenciales sitios terapéuticos son abundantes, sino que hay pruebas de que pueden manipularse de muchas formas diferentes. En lugar de solo activarlos y desactivarlos, podríamos modular su actividad como si fuera un termostato. Desde el punto de vista de la ingeniería, es como si hubiéramos encontrado oro, porque nos da mucho espacio para diseñar «fármacos inteligentes» que se dirigen a lo malo y omiten lo bueno», explica André Faure, investigador postdoctoral del CRG y primer co-autor del artículo.
Las proteínas tienen una función primordial en todos los organismos vivos y desempeñan funciones vitales como aportar estructura, acelerar las reacciones, actuar como mensajeros o combatir enfermedades. Están formadas por aminoácidos y se pliegan en innumerables formas diferentes en el espacio tridimensional. La forma de una proteína es fundamental para su función. Solo un error en una secuencia de aminoácidos puede tener consecuencias potencialmente devastadoras para la salud humana.
La alostería es uno de los grandes misterios sin resolver de la función de las proteínas. Los efectos alostéricos se producen cuando una molécula se une a la superficie de una proteína, lo cual a su vez provoca cambios en un sitio distante en la misma proteína, regulando su función por control remoto. Muchas mutaciones patógenas, incluidos numerosos iniciadores oncogénicos, son patológicas por sus efectos alostéricos.
Pese a su importancia fundamental, los sitios alostéricos son increíblemente difíciles de encontrar, porque las normas que rigen el funcionamiento de las proteínas a nivel atómico no están a la vista. Por ejemplo, una proteína puede cambiar de forma en presencia de una molécula nueva y dejar al descubierto bolsas profundas en su superficie que podrían ser alostéricas pero que no son identificables solo mediante la determinación de estructuras convencional.
Tradicionalmente, los «cazadores de fármacos» han diseñado tratamientos que se dirigen al sitio activo de una proteína, la pequeña región donde se producen las reacciones químicas o donde se unen las dianas. El inconveniente de estos fármacos, conocidos como fármacos ortostéricos, es que los sitios activos de muchas proteínas son muy similares y los fármacos tienden a unirse e inhibir muchas proteínas diferentes a la vez, lo que puede provocar efectos secundarios. En comparación, la especificidad de los sitios alostéricos significa que los fármacos alostéricos son unos de los medicamentos más eficaces que existen en la actualidad. Muchos fármacos alostéricos, que tratan varias afecciones que van desde el cáncer o el sida hasta trastornos hormonales, se han descubierto por casualidad.
Los autores del estudio abordaron este problema desarrollando una técnica llamada PCA de doble profundidad (ddPCA), que describen como un «experimento por fuerza bruta». «Rompemos las cosas adrede de miles de maneras distintas para formar una imagen completa de cómo funciona algo», explica el Profesor de Investigación ICREA Ben Lehner, coordinador del programa Biología de Sistemas del CRG y autor del estudio. «Es como si sospecharas que una bujía no funciona, pero en lugar de comprobar solo eso, el mecánico desmontara todo el coche y revisara todas las piezas una por una. Al analizar diez mil cosas a la vez, identificamos todas las piezas que son realmente importantes».
El método funciona cambiando los aminoácidos que componen una proteína, lo que da lugar a miles de versiones diferentes de la proteína con solo una o dos diferencias en la secuencia. Entonces se analizan todos los efectos de las mutaciones al mismo tiempo en células vivas en el laboratorio.
«Cada célula es una minifábrica que crea una versión diferente de la proteína. En un solo tubo de ensayo tenemos millones de fábricas diferentes y así podemos analizar muy rápidamente cómo funcionan todas las versiones diferentes de una proteína», añade el Dr. Lehner. Los datos obtenidos en los experimentos se introducen en redes neuronales, algoritmos que analizan los datos imitando el funcionamiento del cerebro humano, y esto da lugar a unos mapas completos que determinan la ubicación de los sitios alostéricos en la superficie de las proteínas.
Una de las grandes ventajas del método es que se trata de una técnica asequible, accesible para cualquier laboratorio de investigación del mundo. «Simplifica enormemente el proceso necesario para encontrar sitios alostéricos. La técnica trabaja con un nivel de precisión mejor que varios métodos de laboratorio diferentes más caros y más lentos», comenta Júlia Domingo, primera co-autora del estudio. «Nuestra esperanza es que otros científicos utilicen la técnica para mapear de forma rápida y exhaustiva los sitios alostéricos de las proteínas humanas una a una».
Uno de los beneficios de la técnica a más largo plazo es su potencial para estudiar la función y la evolución de las proteínas. Los autores del estudio creen que, si se amplía, el método podría algún día propiciar avances que puedan predecir con precisión las propiedades de las proteínas a partir de sus secuencias de aminoácidos. Si se lograra, los autores creen que se marcaría el inicio de una nueva era de biología molecular predictiva, que permitiría un desarrollo mucho más rápido de los medicamentos nuevos y una industria limpia y basada en la biología.
«Aunque algunas herramientas pueden predecir la estructura de una proteína leyendo su secuencia, nuestro método va un paso más allá y nos dice cómo funciona una proteína. Forma parte de una visión más amplia en que la biología puede diseñarse mediante ingeniería como los aviones, los puentes o los ordenadores. Llevamos más de 70 años enfrentándonos a los mismos retos, pero resulta que son más manejables de lo que pensábamos. Si lo logramos, se abrirá la puerta a un nuevo campo con posibilidades sin precedentes», concluye el Dr. Lehner.
EN CATALÀ
Una multitud de ‘portes ocultes’ en les proteïnes humanes podrien transformar el descobriment de fàrmacs
El nombre de possibles dianes terapèutiques que hi ha a la superfície de les proteïnes humanes és molt més gran del que es creia, segons els resultats d’un nou estudi que publica la revista Nature.
Una nova tècnica innovadora desenvolupada per un equip científic del Centre de Regulació Genòmica (CRG) de Barcelona ha revelat l’existència de multitud de portes ocultes que controlen la funció de les proteïnes i que, en teoria, podrien utilitzar-se com a diana per canviar dràsticament l’evolució de malalties tan diverses com la demència, el càncer i les malalties infeccioses.
El mètode, en què es duen a terme desenes de milers d’experiments alhora, s’ha utilitzat per traçar el primer mapa d’aquestes dianes tan difícils de trobar, també conegudes com a llocs al·lostèrics, en dues de les proteïnes humanes més habituals, cosa que ha posat de manifest que són abundants i identificables.
La tècnica podria canviar les regles del joc per al descobriment de fàrmacs i donar lloc a uns medicaments més segurs, més intel·ligents i més eficaços. Permet als laboratoris de recerca de tot el món buscar i aprofitar les vulnerabilitats de qualsevol proteïna, fins i tot les que es consideraven «dianes impossibles».
«No és només que aquests potencials llocs terapèutics siguin abundants, sinó que hi ha proves que demostren que es poden manipular de moltes maneres diferents. En lloc de només activar-los i desactivar-los, podríem modular-ne l’activitat com si fos un termòstat. Des del punt de vista de l’enginyeria, és com trobar or, perquè ens dona prou espai per dissenyar “fàrmacs intel·ligents” dirigits al que és dolent i ometen el que és bo», explica André Faure, investigador postdoctoral del CRG i primer co-autor de l’article.
Les proteïnes tenen una funció primordial en tots els organismes vius i duen a terme funcions vitals, com ara aportar estructura, accelerar les reaccions, fer de missatgers o combatre malalties. Estan formades per aminoàcids i es pleguen en innombrables formes diferents en l’espai tridimensional. La forma d’una proteïna és fonamental per a la seva funció. Només un error en una seqüència d’aminoàcids pot provocar conseqüències potencialment devastadores per a la salut humana.
L’al·losteria és un dels grans misteris sense resoldre de la funció de les proteïnes. Els efectes al·lostèrics es produeixen quan una molècula s’uneix a la superfície d’una proteïna, que alhora provoca canvis en un lloc distant de la mateixa proteïna i en regula la funció per control remot. Moltes mutacions patògenes, inclosos nombrosos iniciadors oncogènics, són patològiques pels seus efectes al·lostèrics.
Malgrat la importància fonamental que tenen, els llocs al·lostèrics són increïblement difícils de trobar, ja que les regles que regeixen el funcionament de les proteïnes a nivell atòmic no estan a la vista. Per exemple, una proteïna pot canviar de forma en presència d’una molècula nova i deixar al descobert bosses ocultes profundes a la superfície que són potencialment al·lostèriques però no identificables fent servir només la determinació d’estructures convencional.
Tradicionalment, els «caçadors de fàrmacs» han dissenyat tractaments que es dirigeixen al lloc actiu d’una proteïna, la petita regió on es produeixen les reaccions químiques o on s’uneixen les dianes. L’inconvenient d’aquests fàrmacs, que també es coneixen com a fàrmacs ortostèrics, és que els llocs actius de moltes proteïnes són molt similars i, per tant, els fàrmacs tendeixen a unir-se i inhibir moltes proteïnes diferents alhora, i això pot produir efectes secundaris. En comparació, l’especificitat dels llocs al·lostèrics significa que els fàrmacs al·lostèrics són alguns dels medicaments més eficaços que hi ha actualment. Molts fàrmacs al·lostèrics, que tracten malalties diverses que van des del càncer o la sida fins a trastorns hormonals, s’han descobert per casualitat.
Els autors de l’estudi van abordar aquest problema desenvolupant una tècnica anomenada PCA de doble profunditat (ddPCA), que descriuen com un «experiment per força bruta». «Trenquem les coses expressament de milers de maneres diferents per construir una imatge completa de com funciona alguna cosa», explica el Professor d’Investigació ICREA Ben Lehner, coordinador del programa de Biologia de Sistemes del CRG i autor de l’estudi. «És com si sospites que falla una bugia, però en lloc de comprovar això, el mecànic desmunta tot el cotxe i revisa totes les peces una per una. Analitzant deu mil coses alhora, identifiquem totes les peces que són realment importants.»
El mètode funciona canviant els aminoàcids que formen una proteïna, que dona lloc a milers de versions diferents de la proteïna amb només una o dues diferències en la seqüència. Aleshores s’analitzen tots els efectes de les mutacions alhora en cèl·lules vives al laboratori.
«Cada cèl·lula és una minifàbrica que fa una versió diferent de la proteïna. En un sol tub d’assaig tenim milions de fàbriques diferents i així podem analitzar molt ràpidament com funcionen totes les diferents versions d’una proteïna», afegeix el Dr. Lehner. Les dades obtingudes dels experiments s’introdueixen en xarxes neuronals, algoritmes que analitzen les dades imitant el funcionament del cervell humà, i creen mapes complets que determinen la ubicació dels llocs al·lostèrics a la superfície de les proteïnes.
Un dels grans avantatges del mètode és que és una tècnica assequible, accessible per a qualsevol laboratori de recerca del món. «Simplifica enormement el procés necessari per trobar llocs al·lostèrics. La tècnica funciona amb un nivell de precisió millor que diversos mètodes de laboratori diferents més cars i més lents», explica Júlia Domingo, primera co-autora de l’estudi. «La nostra esperança és que altres científics utilitzin la tècnica per mapar de forma ràpida i exhaustiva els llocs al·lostèrics de les proteïnes humanes una per una.»
Un dels beneficis de la tècnica a més llarg termini és el seu potencial per estudiar la funció i l’evolució de les proteïnes. Els autors de l’estudi creuen que, si s’amplia, el mètode pot algun dia suposar avenços que puguin predir amb precisió les propietats de les proteïnes a partir de les seves seqüències d’aminoàcids. Si s’aconsegueix, els autors creuen que això marcaria l’inici d’una nova era de biologia molecular predictiva, que permetria desenvolupar molt més ràpidament nous medicaments i una indústria neta i basada en la biologia.
«Si bé certs instruments poden predir l’estructura d’una proteïna llegint-ne la seqüència, el nostre mètode va un pas més enllà i ens diu com funciona una proteïna. Forma part d’una visió més àmplia en què la biologia es pot dissenyar mitjançant enginyeria com els avions, els ponts o els ordinadors. Fa més de 70 anys que ens enfrontem als mateixos reptes, però resulta que són més manejables del que ens pensàvem. Si ho aconseguim, s’obrirà la porta a un nou camp amb possibilitats sense precedents», conclou el Dr. Lehner.